La plateforme Migale, dans le cadre de son cycle de formation "Bioinformatique par la pratique" 2022, propose une formation intitulée "Introduction au text-mining avec AlvisNLP" en présentiel à Jouy-en-Josas les 2 et 3 juin 2022. Cette formation s’adresse aux (bio)informaticiens qui souhaitent acquérir des compétences théoriques et pratiques sur l'analyse de données textuelles. La formation abordera les méthodes de Reconnaissance D'Entités Nommées (REN) par des séances théoriques et pratiques.
 Migale Bioinformatics Facility
Migale Bioinformatics Facility
We provide several services for scientists to deal with life sciences data
Our Services
Opening of a system administrator permanent position
A permanent position of "system and network administrator" is open to the 2022 competition within the Migale bioinformatics facility.
Profile description (IE22-MICA-1): https://jobs.inrae.fr/concours/concours-externes-ingenieurs-cadres-techniciens-h-f/ie22-mica-1
Closing date: March 24, 2022.
Nouveau cluster opérationnel / New cluster online
[Update 07.02.22 : bigmem node and queue added]
[Update 16.02.22 : nodes on short, long and infinit.q queue added]
[Update 21.02.22 : highmem.q queue added]
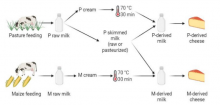
New article: Integration of Multiomic Data to Characterize the Influence of Milk Fat Composition on Cantal-Type Cheese Microbiota
This work is the result of a collaboration with the UMR 0545 of INRAE Aurillac. It can be accessed here
Évolution de l’infrastructure de calcul et stockage / Computing Infrastructure Evolution
Bonjour,
(English below)
Comme annoncé précédemment nous faisons évoluer notre infrastructure informatique en ce début d’année.
Pour les plus pressés d’entre vous, voici les éléments essentiels :
Interruption des accès en ligne de commande et de RSstudio jeudi 3 février de 8h à 10h.
-
Le cluster de calcul est renouvelé de moitié avec des nœuds de calcul plus récents.
Des nouvelles de la plateforme Migale pour ce début d’année / News of Migale in the beginning of the year
Bonjour,
(english below)
Il reste encore quelques jours pour vous souhaiter à toutes et tous une très bonne année 2022.
La crise sanitaire a de nouveau impacté nos activités mais nous avons su en 2021, comme l’année précédente, adapter notre offre de service aux contraintes. Nous avons pu, par exemple, maintenir l’ensemble des modules du cycle de formation 2021, en en basculant certains à distance.
"Bioinformatics by practicing" cycle 2022
 The 2022 training cycle is open
The 2022 training cycle is open
You can consult the program and the schedule here: https://migale.inrae.fr/trainings
Informations sur l'utilisation de FROGS sous Galaxy
Comme vous le savez, FROGS est un outil en constante évolution. En conséquence, des mises à jour interviennet régulièrement.
Jusqu'à maintenant, les versions de FROGS cohabitaient dans l'interface, ce qui pouvait parfois entrainer des confusions. Depuis peu, Galaxy propose une nouvelle façon de gérer les versions d'outils (pour les outils récemment déployés). Un nouveau bouton apparaît en haut à droite du formulaire, et il est possible de choisir la version de l'outil.
New publication: FROGS: a powerful tool to analyse the diversity of fungi with special management of internal transcribed spacers
A very short newsletter to announce the release of the new FROGS article entitled "FROGS: a powerful tool to analyse the diversity of fungi with special management of internal transcribed spacers", which you can read here : https://academic.oup.com/bib/advance-article/doi/10.1093bib/bbab318/6354026?guestAccessKey=c3c28079-333a-432e-9d9e-b4c37da8284d.
The tick microbiota: its role in the carriage and transmission of pathogens
A nice collaborative work highlighted on the INRAE website