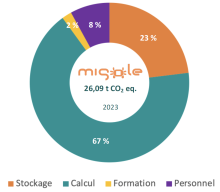
Dans le cadre de sa politique environnementale, et en lien avec la norme ISO 9001, la plateforme Migale a souhaité évaluer le bilan des émissions de gaz à effet de serre de son infrastructure matérielle pour l’année 2023.
 Migale Bioinformatics Facility
Migale Bioinformatics Facility
We provide several services for scientists to deal with life sciences data
Our Services
Migale, one of the Collective Scientific Infrastructure of INRAE, is part of the BioinfOmics Research Infrastructure of INRAE for bioinformatics. It is also a member of IFB (Institut Français de Bioinformatique), the French bioinformatics infrastructure and associated facility of France Génomique, the French genomic infrastructure for which we contribute to support different developments in bioinformatics.
A free account gives you access to work and save directories for your data, and access to the computer farm for your analyses.
The cluster farm is composed of about a thousand cores organized in different queues. We use the Sun Grid Engine queuing system for managing jobs.
You have a free access to our Galaxy server. Galaxy allows non-bioinformaticians to easily run tools without technical knowledges.
Command line tools, R packages and Galaxy wrappers are available on request and accessible to all migale authenticated users.
We provide an access to a large set of public biological databanks including whole genomes, nucleic and proteic sequences and other resources. They are updated automatically with BioMaJ or upon request.
We write tutorials to help you get familiar with tools, best practices, languages, etc.
Each year, we offer our "Bioinformatics by practicing" cycle. This cycle covers a broad spectrum of bioinformatics. The modules mix theoretical part and practical work.
We answer to the most common questions regarding the technical difficulties you can go through on our infrastructure.
Find all the ways to contact us.
Update of our infrastructure
Cette actualité sera mise à jour régulièrement pour vous informer de l'inclusion des nouveau noeuds
This news item will be updated continuously to reflect the inclusion of new nodes
Comme annoncé précédemment, nous avons basculé cet après-midi sur le nouveau cluster de calcul.
Ce qui ne change pas :
Online command-line and Rstudio access unavailable from Tuesday, April 22 at 3 PM until Wednesday, April 23 at 12 PM
Update of our infrastructure
This spring we are upgrading our IT infrastructure.